江千涛
作者: 审稿人: 时间: 2018-04-08 点击次数:次

江千涛,博士,教授
Qiantao Jiang Ph.D, Professor
Email:
qiantaojiang@sicau.edu.cn
qiantaojiang@qq.com
个人简介:
四川农业大学1998级农学学士、2002级作物遗传育种硕士、2004级四川农业大学-加拿大农业部联合培养博士。国际小麦品质改良专家组成员,四川省学术和技术带头人后备人选,四川省杰出青年培育基金获得者,四川省海外留学人员。以第一或通讯作者在Plant Biotechnology Journal、Carbohydrate Polymers、Food Chemistry、Theretical and Applied Genetics等SCI期刊发表论文50余篇,出版Springer基因编辑专著章节;主持国家自然科学基金、转基因专项子课题、四川省学术技术带头人培育项目等国家和省部级项目。第一发明人获发明专利授权5项;主研选育小麦新品种10个。主研获四川省科技进步奖一等奖2项、二等奖1项。
博士&硕士招生专业:作物遗传育种,农艺与种业
个人主页Researchgate website: https://www.researchgate.net/profile/Qian-Tao_Jiang?ev=prf_highl
研究方向:
1. 麦类遗传转化和基因编辑
建立了成熟、高效的农杆菌介导的麦类作物(小麦和大麦)转基因和基因编辑技术体系,通过不断探索初步建立了不受基因型限制的小麦转基因体系,已在“蜀麦”系列品种中成功进行转化。通过单个或多个基因过表达和定向敲除,开展淀粉、蛋白等籽粒组分遗传修饰研究。
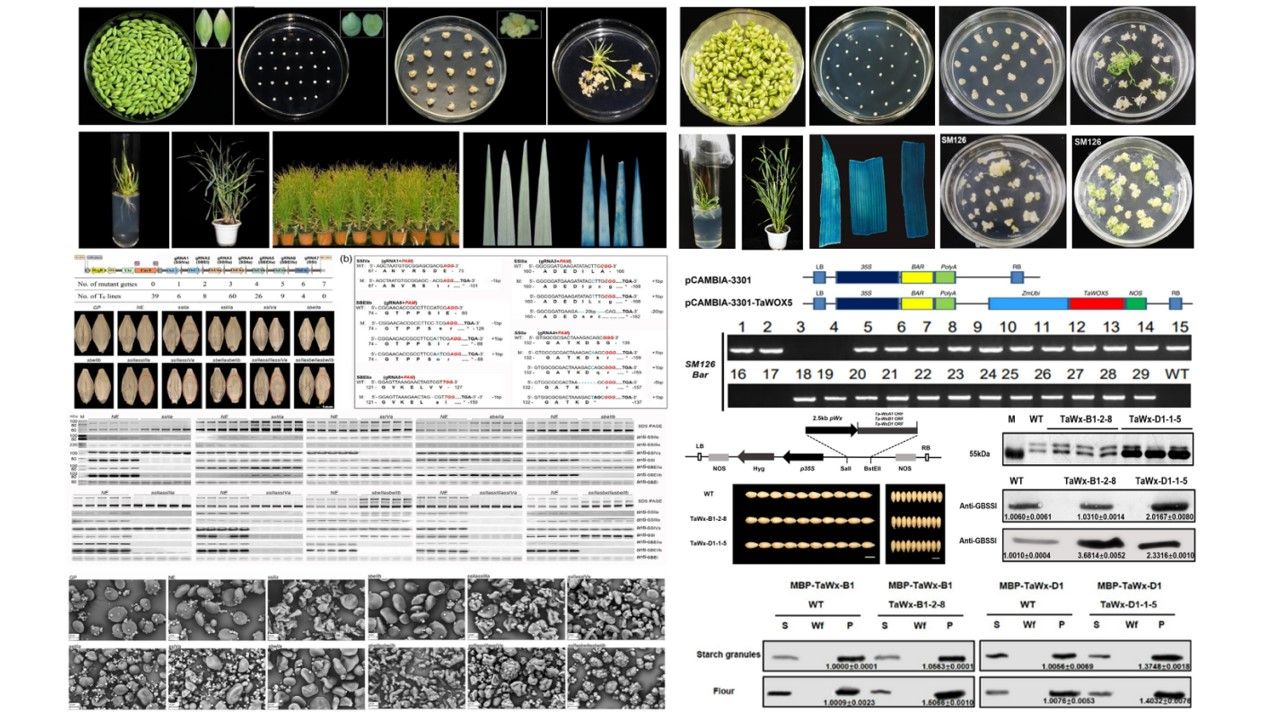
2. 麦类淀粉代谢调控与籽粒发育研究
利用组学和分子生物学技术,挖掘调控淀粉合成、微观结构和组成的关键基因,解析淀粉合成调控的关键基因。聚集籽粒灌浆发育过程,揭示淀粉合成响应籽灌浆发育调控网络和分子机制。
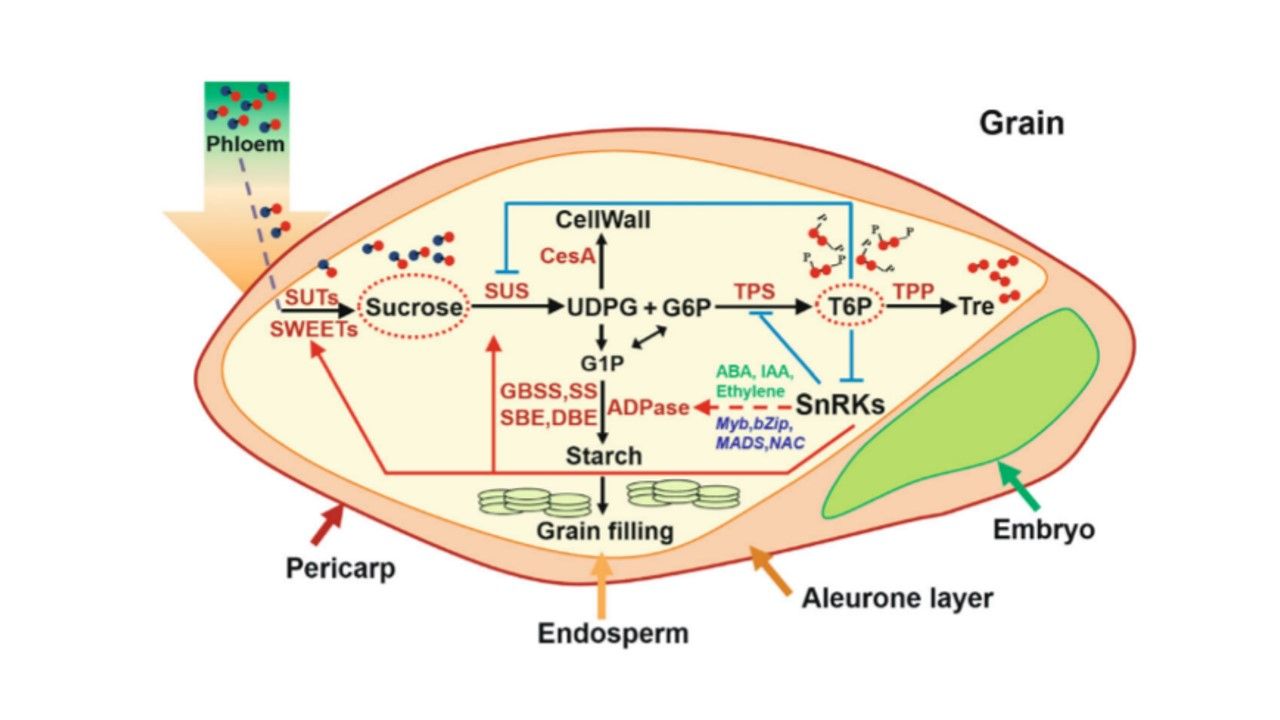
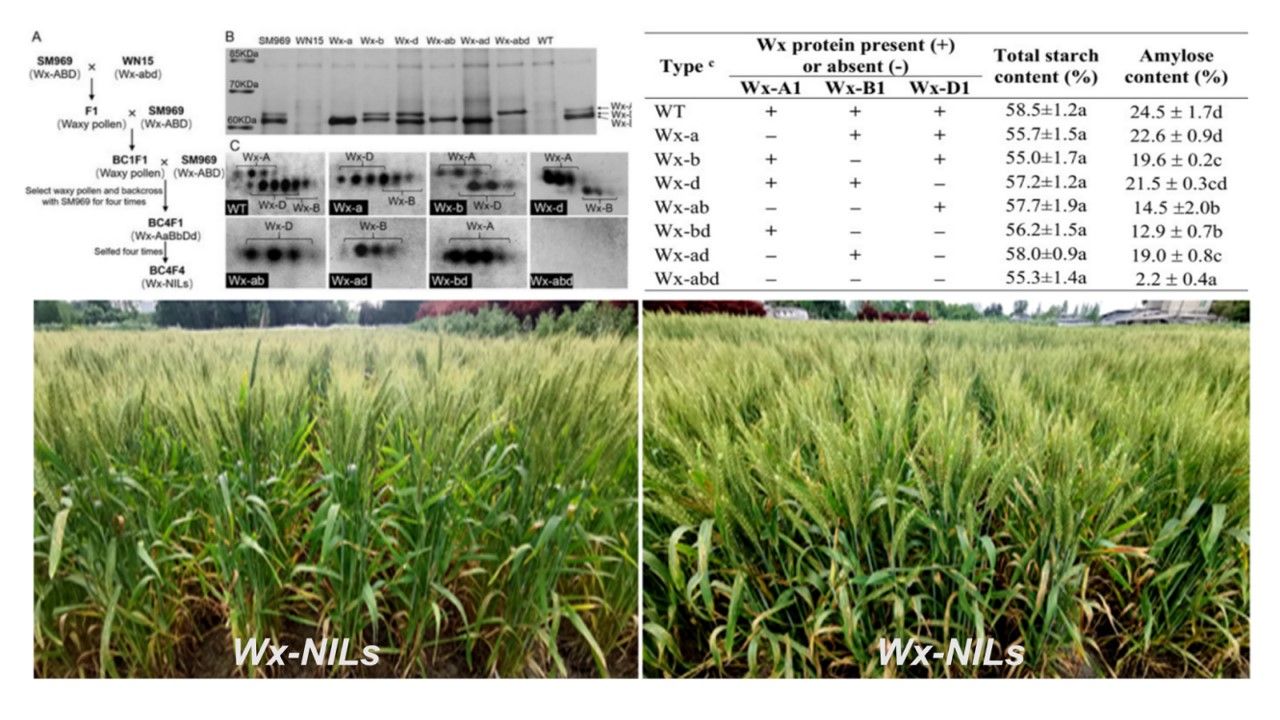
3. 特异淀粉麦类新种质创制
利用基因编辑和化学诱变对淀粉关键基因进行定向突变,创制高抗性淀粉、高支链(糯性)淀粉、高糖分含量的特异麦类作物新种质,以满足降血糖、预防肠道疾病、面食制品和酿酒等不同营养和加工品质需求。
国际交流合作:
担任四川省国际科技合作基地“小麦遗传育种联合研究中心”负责人,与国际知名机构建立了广泛的合作关系。
外方合作单位:
英国约翰因纳斯中心(John Innes Centre,JIC),Dr. David Seung,开展小麦淀粉颗粒形成机制合作,联合培养博士1名。
西班牙科尔多瓦大学(Universidad de Córdoba),Dr. Carlos Guzmán García高级科学家,开展大麦淀粉合成调控研究,获四川省国际科技创新项目(2022YFH0055)资助,合作发表论文6篇。
澳大利亚联邦科工组织(CSIRO),Dr. Zhongyi Li高级科学家,开展淀粉精细表型合作,联合培养博士1名,合作发表论文6篇。
英国约翰因纳斯中心(John Innes Centre,JIC),Dr.Wendy Harwood教授,开展大麦遗传转化和基因编辑合作,获四川省国际科技创新项目(2020YFH0140)资助,联合培养博士1名,合作发表论文9篇,撰写大麦基因编辑技术指南丛书章节1部。
加拿大农业与农业食品部(Agriculture and Agri-Food Canada,AAFC),Dr. Zhenxiang Lu高级科学家,开展特异淀粉分子机制合作, 获四川省国际交流合作项目(2016HH0057)资助,联合培养博士1名,合作发表论文16篇。
国际交流访问:
v 2023年6月,赴西班牙马德里参加第14届国际谷蛋白大会,并在做题为“Characterization of novel HMW glutenin subunits from Aegilops sharonensis which improves bread- making quality”的大会学术报告;受邀担任Expert Working Group (EWG) on Improving Wheat Quality for Processing And Health国际小麦加工和健康品质改良项目专家组成员;受邀赴科尔多瓦大学Carlos Guzman教授进行合作交流。
v 2019年12月,邀请澳大利亚Zhongyi Li博士,国际玉米小麦改良中心(现为西班牙卡尔多瓦大学研究员)Carlos Guzman博士来华进行访问,通过学术报告,专题讲授和实验技术培训等形式开展学术交流。
v 2019年7月,参加第14届国际谷物穗发芽大会,并受邀作大会报告“Modification to starch of wheat and barle y by chemical mutagenesis and genome edition”。
v 2019年7月,赴加拿大萨斯卡通参加第一届国际小麦大会。
v 2019年6月,受英国生物技术和生物科学研究理事会(BBSRC)国际合作项目的资助,访问了洛桑研究所(Rothamsted Research),与麦类品质研究世界知名专家Peter Shewry进行学术交流。
v 2018年3月,应国际玉米小麦改良中心(CIMMTY)麦类淀粉研究知名学者,第九届国际谷蛋白大会组委会主席,Carlos Guzman博士邀请,赴墨西哥参加该次大会。
v 2017年4月,赴奥地利维也纳参加第13届国际小麦遗传大会。
v 2016年1月,赴澳大利亚联邦科工组织(CSIRO)访问,与CSIRO Agriculture and Food分部的知名淀粉研究专家Zhongyi Li博士就合作研究工作进行商讨。
v 2013年9月,赴日本横滨参加第12届国际小麦遗传大会。
v 2018年10月,邀请澳大利亚CSIRO高级科学家Zhongyi Li博士来华交流访问。
v 2008年1月-2009年1月,受国家留学基金委资助,在加拿大农业部列桥研究中心高级科学家Lu Zhen-Xiang实验室,从事小黑麦淀粉合成调控研究。

在读留学生:
Hassan Karim,作物遗传育种,博士,2021级,巴基斯坦
Sujon Kumar,作物遗传育种,博士,2019级,孟加拉国
社会服务:
担任四川省农业重大技术协同推广岗位专家、成都市农业科技专家服务团成员、崇州市小春技术服务团团长,在崇州、大邑、广汉、大英等全省不同生态点开展小麦新品种示范推广,助力高质量天府粮仓建设和保障四川粮食安全。

2023年4月6日中共四川省委书记王晓辉视察小麦示范推广工作
代表论文:
v Yang Q, Ral JP, Wei Y, Zheng Y, Li Z, Jiang Q*. Genome editing of five starch synthesis genes produces highly resistant starch and dietary fibre in barley grains. Plant Biotechnol J. 2024 Jul;22(7):2051-2053
v Hu Q, Liu J, Chen X, Guzmán C, Xu Q, Zhang Y, Chen Q, Tang H, Qi P, Deng M, Ma J, Chen G, Wei Y, Wang J, Zheng Y, Tu Y, Jiang Q*. Multi-omic analysis reveals the effects of interspecific hybridization on the synthesis of seed reserve polymers in a Triticum turgidum ssp. durum × Aegilops sharonensis amphidiploid. BMC Genomics. 2024 Jun 20;25(1):626
v Li Y, Li Q, Hu Q, Guzman C, Lin N, Xu Q, Zhang Y, Lan J, Tang H, Qi P, Deng M, Ma J, Wang J, Chen G, Lan X, Wei Y, Zheng Y, Jiang Q*. Aegilops sharonensis HMW-GSs with unusually large molecular weight improves bread-making quality in wheat-Ae. sharonensis introgression lines. J Sci Food Agric. 2023 Mar 15;103(4):1668-1675
v Jiang Q*, Yang Q, Harwood W, Tang H, Wei Y, Zheng Y. A CRISPR/Cas9 Protocol for Target Gene Editing in Barley. Methods Mol Biol. 2023;2653:287-296
v Karim H, Kumar S, Lan J, Tang H, Guzmán C, Xu Q, Zhang Y, Qi P, Deng M, Ma J, Wang J, Chen G, Lan X, Wei Y, Zheng Y, Jiang Q*. Analysis of starch structure and functional properties of tetraploid wheat (Triticum turgidum L.) with differing waxy protein composition. J Sci Food Agric. 2022 Oct;102(13):5974-5983
v Feng X, Rahman MM, Hu Q, Wang B, Karim H, Guzmán C, Harwood W, Xu Q, Zhang Y, Tang H, Jiang Y, Qi P, Deng M, Ma J, Lan J, Wang J, Chen G, Lan X, Wei Y, Zheng Y, Jiang Q*. HvGBSSI mutation at the splicing receptor site affected RNA splicing and decreased amylose content in barley. Front Plant Sci. 2022 Sep 23;13:1003333
v Li Y, Karim H, Wang B, Guzmán C, Harwood W, Xu Q, Zhang Y, Tang H, Jiang Y, Qi P, Deng M, Ma J, Lan J, Wang J, Chen G, Lan X, Wei Y, Zheng Y, Jiang Q*. Regulation of Amylose Content by Single Mutations at an Active Site in the Wx-B1 Gene in a Tetraploid Wheat Mutant. Int J Mol Sci. 2022 Jul 29;23(15):8432
v Yang Q, Ding J, Feng X, Zhong X, Lan J, Tang H, Harwood W, Li Z, Guzmán C, Xu Q, Zhang Y, Jiang Y, Qi P, Deng M, Ma J, Wang J, Chen G, Lan X, Wei Y, Zheng Y, Jiang Q*. Editing of the starch synthase IIa gene led to transcriptomic and metabolomic changes and high amylose starch in barley. Carbohydr Polym. 2022 Jun 1;285:119238
v Zhang X, Karim H, Feng X, Lan J, Tang H, Guzmán C, Xu Q, Zhang Y, Qi P, Deng M, Ma J, Wang J, Chen G, Lan X, Wei Y, Zheng Y, Jiang Q*. A single base change at exon of Wx-A1 caused gene inactivation and starch properties modified in a wheat EMS mutant line. J Sci Food Agric. 2022 Mar 30;102(5):2012-2022
v Ding J, Karim H, Li Y, Harwood W, Guzmán C, Lin N, Xu Q, Zhang Y, Tang H, Jiang Y, Qi P, Deng M, Ma J, Wang J, Chen G, Lan X, Wei Y, Zheng Y, Jiang Q*. Re- examination of the APETALA2/Ethylene-Responsive Factor Gene Family in Barley (Hordeum vulgare L.) Indicates a Role in the Regulation of Starch Synthesis. Front Plant Sci. 2021 Dec 1;12:791584
v Zhong X, Feng X, Li Y, Guzmán C, Lin N, Xu Q, Zhang Y, Tang H, Qi P, Deng M, Ma J, Wang J, Chen G, Lan X, Wei Y, Zheng Y, Jiang Q*. Genome-wide identification of bZIP transcription factor genes related to starch synthesis in barley (Hordeum vulgare L.). Genome. 2021 Dec;64(12):1067-1080. doi: 10.1139/gen-2020-0195. Epub 2021 May 31. PMID: 34058097.
v Zhong X, Lin N, Ding J, Yang Q, Lan J, Tang H, Qi P, Deng M, Ma J, Wang J, Chen G, Lan X, Wei Y, Zheng Y, Jiang Q*. Genome-wide transcriptome profiling indicates the putative mechanism underlying enhanced grain size in a wheat mutant. 3 Biotech. 2021 Feb;11(2):54
v Yang Q, Zhong X, Li Q, Lan J, Tang H, Qi P, Ma J, Wang J, Chen G, Pu Z, Li W, Lan X, Deng M, Harwood W, Li Z, Wei Y, Zheng Y, Jiang Q*. Mutation of the d-hordein gene by RNA-guided Cas9 targeted editing reducing the grain size and changing grain compositions in barley. Food Chem. 2020 May 1;311:125892
v Li X, Li Y, Karim H, Li Y, Zhong X, Tang H, Qi P, Ma J, Wang J, Chen G, Pu Z, Li W, Tang Z, Lan X, Deng M, Li Z, Harwood W, Wei Y, Zheng Y, Jiang Q*. The production of wheat - Aegilops sharonensis 1Ssh chromosome substitution lines harboring alien novel high-molecular-weight glutenin subunits. Genome. 2020 Mar;63(3):155-167
v Luo M, Ding J, Li Y, Tang H, Qi P, Ma J, Wang J, Chen G, Pu Z, Li W, Li Z, Harwood W, Lan X, Deng M, Lu Z, Wei Y, Zheng Y, Jiang Q*. A single-base change at a splice site in Wx-A1 caused incorrect RNA splicing and gene inactivation in a wheat EMS mutant line. Theor Appl Genet. 2019 Jul;132(7):2097-2109
v Yang Q, Li S, Li X, Ma J, Wang J, Qi P, Chen G, Pu Z, Li W, Harwood W, Li Z, Liu BL, Lan X, Deng M, Lu Z, Wei Y, Zheng Y, Jiang Q*. Expression of the high molecular weight glutenin 1Ay gene from Triticum urartu in barley. Transgenic Res. 2019 Apr;28(2):225-235
v Zhou YJ, Yang Q, Zhong XJ, Tang HP, Deng M, Ma J, Qi PF, Wang JR, Chen GY, Liu YX, Lu ZX, Li W, Lan XJ, Wei YM, Zheng YL, Jiang QT*. Alternative splicing results in a lack of starch synthase IIa-D in Chinese wheat landrace. Genome. 2018 Mar;61(3):201-208
v Zhang LL, Chen H, Luo M, Zhang XW, Deng M, Ma J, Qi PF, Wang JR, Chen GY, Liu YX, Pu ZE, Li W, Lan XJ, Wei YM, Zheng YL, Jiang QT*. Transposon insertion resulted in the silencing of Wx-B1n in Chinese wheat landraces. Theor Appl Genet. 2017 Jun;130(6):1321-1330
v Jiang QT*, Zhang XW, Ma J, Wei L, Zhao S, Zhao QZ, Qi PF, Lu ZX, Zheng YL, Wei YM. Characterization of high-molecular-weight glutenin subunits from Eremopyrum bonaepartis and identification of a novel variant with unusual high molecular weight and altered cysteine residues. Planta. 2014 Apr;239(4):865-75
v Jiang QT*, Zhao QZ, Wang XY, Wang CS, Zhao S, Cao X, Lan XJ, Lu ZX, Zheng YL, Wei YM. Characterization of x-type high-molecular-weight glutenin promoters (x-HGP) from different genomes in Triticeae. Springerplus. 2013 Apr 10;2(1):152
v Jiang QT*, Ma J, Zhao S, Zhao QZ, Lan XJ, Dai SF, Lu ZX, Zheng YL, Wei YM. Characterization of HMW-GSs and their gene inaction in tetraploid wheat. Genetica. 2012 Sep;140(7-9):325-35
v Jiang QT*, Ma J, Wei YM, Liu YX, Lan XJ, Dai SF, Lu ZX, Zhao S, Zhao QZ, Zheng YL. Novel variants of HMW glutenin subunits from Aegilops section Sitopsis species in relation to evolution and wheat breeding. BMC Plant Biol. 2012 May 30;12:73. doi: 10.1186/1471-2229-12-73
v Jiang QT*, Liu T, Ma J, Wei YM, Lu ZX, Lan XJ, Dai SF, Zheng YL. Characterization of barley Prp1 gene and its expression during seed development and under abiotic stress. Genetica. 2011 Oct;139(10):1283-92
v Jiang QT*, Wei YM, Lu ZX, Pu ZE, Lan XJ, Zheng YL. Structural variation and evolutionary relationship of novel HMW glutenin subunits from Elymus glaucus. Hereditas. 2010 Jun 1;147(3):136-41
v Jiang QT*, Wei YM, Wang F, Wang JR, Yan ZH, Zheng YL. Characterization and comparative analysis of HMW glutenin 1Ay alleles with differential expressions. BMC Plant Biol. 2009 Feb 6;9:16


