利用两个基因组已知的亲本构建群体定位大麦控制粒重的关键QTL位点并预测候选基因
作者: 审稿人:刘宇娇 时间: 2020-12-25 点击次数:次
粒重和单位面积穗粒数是影响大麦产量的主要因素。粒重主要受遗传因素影响,同时也是广义遗传率较高、性状遗传较稳定的产量构成因子。在高产栽培条件下,由于单位面积穗粒数受到限制,粒重成为影响产量的最主要因素。因此,了解粒重的遗传基础对于进一步提高大麦产量尤为重要。然而,大麦基因组比水稻、玉米更加复杂,粒重基因的精细定位、克隆及功能研究进展相对缓慢,现阶段国内外研究主要围绕籽粒基因的初定位及其遗传效应展开,并已在大麦7条染色体上鉴定到多个QTL位点,但是均没有更进一步的研究。因此,解析大麦粒重的遗传机制,是提高大麦产量的一个有效策略。
近日四川农业大学小麦研究所和澳大利亚CSIRO Agriculture Flagship在The Crop Journal上发表了题为“Identification of loci and candidate genes controlling kernel weight in barley based on a population for which whole genome assemblies are available for both parents”的论文。该论文以小粒重野生大麦AWCS276为共同亲本,分别和大粒重栽培大麦Morex和Baudin构建了两个重组自交系(MA和BA)(图1),并对MA群体进行了genotyping by sequence (GBS)测序构建高密度遗传连锁图谱,在多环境和多群体对百粒重位点进行了挖掘和鉴定。
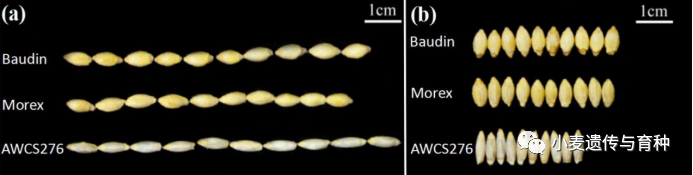
图1 大粒重栽培大麦(Morex和Baudin)和小粒重野生大麦AWCS276籽粒图
结果
1.百粒重QTL检测
在BA群体里一共鉴定到5个QTL控制百粒重,分布在2H、3H、3H、6H和7H,可解释的表型变异在10.5 (KW-BA-2H)和30.3% (KW-BA-3H.2) 之间。其中只有KW-BA-6H能在连续3年所有环境下鉴定到,LOD值在3.8和5.8之间。同样在MA群体里鉴定到5个QTL,分布在2H、3H、6H、7H和7H,其中两个QTL(KW-MA-2H和KW-MA-6H)能在所有4个环境下鉴定到。最后在两个群体多个环境下鉴定得到3个主效QTL位点,分别位于2HL、6HL和7HL上(图2)。
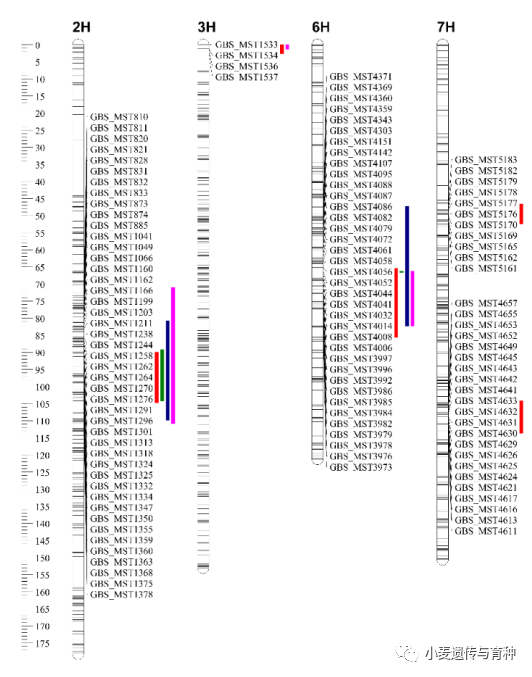
图2 MA群体中控制粒重的QTL分布图
2.百粒重主效QTL位点KW-6HNILs创制
在3个主效位点中,位于6HL上的位点KW-6H能在两个群体的所有环境下检测到,因此针对该位点构建了近等基因系(NILs)。将筛选得到的9对NILs种到田间和温室,表型调查发现,其中7对NILs的粒重存在显著差异,而其他农艺性状均没有显著差异。
3.百粒重主效QTL位点候选基因预测
研究者利用已公布的MA群体两个亲本(Morex和AWCS276)参考基因组和水稻粒重相关同源基因序列,对主效QTL位点进行候选基因预测。KW-2H和KW-6H位点分别预测到3个和1个候选基因,通过亲本序列比对发现,4个候选基因均存在基因序列的SNP或者Indel差异(图3)。以上结果为后续精细定位和克隆大麦粒重QTL奠定了基础,同时证明利用已知基因组序列亲本构建的遗传群体进行目标性状的遗传定位,有利于对候选基因的预测挖掘。

图3KW-2H和KW-6H的候选基因的基因结构和在亲本(Morex和AWCS276)之间的序列差异
讨论
本研究中两个重组自交系的共同亲本AWCS276是中东野生大麦,没有经过人为选择,驯化程度较低,含有栽培大麦没有的优异基因,可为遗传变异和作物改良提供丰富的种质资源,已经广泛的被用于大麦遗传育种。而另外两个亲本Morex和Baudin是高产栽培品种,他们与AWCS276均有较远的血缘关系,且百粒重具有较大的差异,所以由野生大麦AWCS276和栽培大麦品种Morex、Baudin构建的遗传群体具有丰富的遗传基础,有利于野生大麦和栽培大麦间百粒重的QTL定位。同时栽培大麦Morex和野生大麦AWCS276已先后于2017年和2020年公布了参考基因组数据。两个亲本材料的基因组数据也为后续主效QTL位点的候选基因的挖掘提供了序列基础。本研究利用两个重组自交系群体鉴定到10个控制百粒重的QTL,这些位点在前人研究中均有报道,说明这些QTL在不同的遗传背景和环境下较稳定。同时通过比对这些QTL位点的物理位置发现它们均位于染色体的长臂上。众所周知,不同的群体里的重组率可能有所不同,但依然不能解释本研究发现的所有来自野生大麦的主效位点都位于染色体长臂且大部分都靠近端粒是否只是巧合。
本文鉴定到主效QTL位点HKW-6H能在两个群体的所有环境下检测到,因此针对该位点用HIF法构建了近等基因系。在MA群体筛选得到的9对NILs,其中7对NILs的百粒重存在显著差异,而其他性状均没有显著差异。然而对水稻粒重基因的研究发现,许多农艺性状都与粒重相互作用,如籽粒长宽、分蘖、旗叶生长。而HKW-6H的NILs之间只有百粒重有显著差异,表明在单个基因型中控制粒重的位点很小,而针对较小的位点的鉴定和深入挖掘要比继续检测新的位点更有利于大麦育种工作。
HIF方法构建NILs最初是利用侧翼标记去筛选杂合植株。如果两个侧翼标记都与它们的目标位点紧密相连,这种方法的成功率较高。然而,当使用来自QTL定位的侧翼标记与目标位点距离较远时,连锁阻力可能是一个重要的问题。因为一对NILs之间的最小差异会是两个侧翼标记之间的染色体片段,所以当用QTL初定位结果的侧翼标记构建NILs时,如果侧翼标记与目标基因座没有紧密连锁,NILs之间差异片段可能会很大。为了获得一对NILs之间差异的染色体片段较小的NILs,可以只使用一个与目标位点紧密连锁的标记去构建NILs。然而,一个标记和它的目标基因座之间也可能发生重组,因此使用单个标记获得的一些NILs可能是错误的,它们不会在目标位点分离。这可能是在本研究中构建的9对NILs中有两对在粒重上没有差异的原因,这也与前人研究中获得的结果相似。
周红、罗伟、高尚为本文共同第一作者,刘春吉和刘亚西为共同通讯作者。该研究得到了国家自然科学基金项目(31771794),国家重点研发计划项目(2017YFD0100900、2016YFD0101004),四川省科技厅青年基金项目(2016JQ0040),科技部国际合作专项项目(2015DFA306002015-GH03-00008-HZ)的资助。
转载来源:小麦研究联盟
原文链接:https://doi.org/10.1016/j.cj.2020.07.010


